Martin Obermaier, Berlin
Neue Methoden für Neisseria gonorrhoeae, Mycoplasma genitalium und Chlamydia trachomatis
Zum Nachweis einer Infektion mit Neisseria gonorrhoeae (NG), Mycoplasma genitalium (MG) und Chlamydia trachomatis (CT) werden derzeit in erster Linie Nukleinsäure Amplifikationstechniken (NAAT, Realtime-PCR, TMA oder ähnliches) durchgeführt. Diese Verfahren haben den Vorteil einer raschen und empfindlichen Erregerdiagnostik. Dem gegenüber stehen deutliche Lücken in der Beurteilung der Antibiotika-Empfindlichkeit. Die üblicherweise verwendeten Transportmedien für NAAT, die bei Raumtemperatur Nukleinsäure-stabilisierend wirken und somit eine deutliche Verbesserung der Empfindlichkeit ermöglichen, erreichen dies durch Denaturierung von Proteinen und Lyse von Viren, Zellen und Bakterien. Eine Anzucht der Keime zur phänotypischen Resistenzbestimmung mittels Wachstumsbestimmung bei unterschiedlichen Antibiotikakonzentrationen ist aus diesem Material nicht möglich.

Martin Obermaier im Labo
Alternative Abstrichmaterialien, die sowohl NAAT als auch eine Anzucht ermöglichen, müssen von dem durchführenden Labor erst validiert werden und zeigen zudem meist Einschränkungen in der Probenstabilität und Empfindlichkeit der Messung. Die Kultur von NG ist nur mit Spezialmedien und nur bei großer Erregermenge möglich, mit einem deutlich besseren Anzuchterfolg, wenn die Probe innerhalb von zwei Stunden weiterverarbeitet wird. Zahlreiche spezielle Transportmedien sollen den Anzuchterfolg verbessern und sind sicher ein relevanter Baustein, können aber schnelle Bearbeitung, korrekte Lagerung und Transport bei Raumtemperatur nicht ersetzen. Bei optimierten Maßnahmen werden durchaus gute Anzuchterfolge erzielt. Zum Beispiel können aus Urethralabstrichen bei direkter Verarbeitung nach Abnahme (bedside inoculation) bis zu 93% der positiven Proben symptomatischer Patienten angezüchtet werden. Bei asymptomatischen Personen liegt die Erfolgsrate aber nur noch bei 45%.1
Spezielle Medien
Trotz dieser Schwierigkeiten wirkt die Anzucht von NG trivial im Vergleich zu MG und CT. Bei MG existieren einzelne Berichte zu gelungener Anzucht mittels spezieller Nährmedien oder mittels Zellkultur. Die Erfolgsraten in der Zellkultur sind besser als im zellfreien Nährmedium, dauern aber auch mehrere Wochen bei nur 60% Erfolgsrate selbst in Proben mit hoher Bakterienlast.2 Ähnliche Erfolgsraten zeigen sich bei CT mit dem Unterschied einer schnelleren Anzucht.3,4
Während für NG eine phänotypische Resistenztestung grundsätzlich möglich und die erforderliche Ausrüstung in vielen mikrobiologischen Laboren vorhanden ist, ist dies bei CT und MG nur in spezialisierten Referenzlaboren der Fall. Ein weiteres Problem: Die Vergütung der Resistenztestung von NG mittels Anzucht durch die Krankenversicherung ist nicht ausreichend, da es keine automatisierten Verfahren gibt.
Neisseria gonorrhoeae
Antimikrobielle Resistenz stellt bei NG ein zunehmendes Problem dar. Surveillance und Monitoring sind dringend erforderlich und werden in Deutschland vom Robert Koch-Institut auch durchgeführt. Aufgrund der oben genannten Komplexität findet dies aber nicht in ausreichendem Maße statt. Eine über Resistenztestung mittels Anzucht-gesteuerte antibiotische Therapie ist zeitlich nicht sinnvoll zu realisieren. Molekulardiagnostische Methoden können das Diagnosefenster verkleinern und Kulturprobleme lösen, zeigen neben ihren Stärken aber auch – abhängig vom untersuchten Resistenzmechanismus – Schwächen.
Bei den molekulardiagnostischen NAAT-Methoden unterscheidet man Verfahren, die auf spezifischem Nachweis von Punktmutationen im bakteriellen Genom basieren von Verfahren zur Sequenzierung von Genbereichen oder vollständigen Genomen.
Punktmutationen
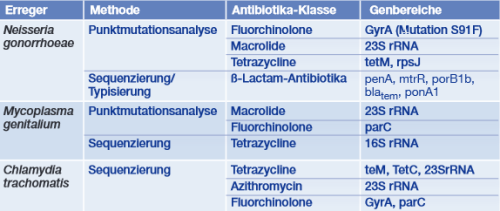
Tab. 1 Molekulardiagnostische Methoden zur Resistenztestung
Methodisch am weitesten etabliert ist die Punktmutationsanalyse der Mutation S91F im gyrA-Gen von NG, welche Ciprofloxacin-Resistenz vermittelt. 99% aller untersuchten Patienten würden abhängig von dem Ergebnis dieser Testung korrekt mit einem Fluorchinolon behandelt werden können.5 Die im Rahmen der Surveillance des Go-Surv-AMR am Robert Koch-Institut durchgeführten Untersuchungen zeigen eine Rate von über 60% Resistenz gegen Ciprofloxacin, was den Stellenwert einer schnellen Testung dieser einen Punktmutation vermindert. Dem gegenüber steht die Möglichkeit einer oral verfügbaren Therapie und der Einsparung von Ceftriaxon in knapp 40% der Fälle. Für die bisher noch nicht verfügbaren Topoisomerase II-Hemmer Zoliflodacin und Gepotidacin, die in den Studien Wirksamkeit gegen NG gezeigt haben, sind die relevanten Mutationen, die mit einer Resistenz assoziiert sind, schon bekannt.6,7
Ebenfalls gut beschrieben sind die Mutationen A2059G und C2611T der bakteriellen ribosomalen RNA, die stark mit einer Resistenz gegen Macrolid-Antibiotika assoziiert sind.8 Resistenz gegen Tetracycline ist assoziiert mit dem Nachweis des tetM Gens, aber auch mit der Mutation V57M im rpsJ-Gen.9 Obwohl publizierte Methoden zum Nachweis vorhanden sind, ist bei über 95% Resistenz gegen Tetracyclin in Deutschland der Nutzen einer Implementierung der Suche nach diesen Mutationen fraglich – insbesondere auch vor dem Hintergrund einer möglicherweise zunehmenden Tetrazyklin-Resistenz unter Doxy-P(r)EP.10
Resistenz gegen Beta-Lactam Antibiotika wird durch verschiedene Gene vermittelt (penA, mtrR, porB1b, blatem-Varianten oder ponA1), die in unterschiedlichem Ausmaß auch die Empfindlichkeit gegen Cephalosporine beeinflussen. Punktmutationsanalysen für die einzelnen Gene sind möglich, aber aufgrund der Anzahl ist hier die Analyse mittels Sequenzierung inzwischen effizienter. Mit den entsprechenden Datenbanken können Sequenztypen identifiziert werden, die entsprechende Resistenzcharakteristiken zeigen. Dies ist nicht nur mittels Sequenzierung der kompletten Genome (whole genome sequencing; bei NG 2.2 Megabasen) möglich, sondern auch, wie bei NG-STAR, durch Sequenzierung der typischen Gene mit den Resistenz-assoziierten Veränderungen.11 Eine Sequenzierung der kompletten Genome ist nur aus Reinkulturen mit akzeptablem Aufwand möglich. Die Typisierung durch Sequenzierung einzelner Genbereiche ist mittels Nanoporen-Sequenzierung inzwischen mit vertretbarem wirtschaftlichem und zeitlichem Aufwand möglich. Hoch resistente Stämme, wie der Sequenz-Typ 16406 mit ausgeprägter Resistenz gegen Ceftriaxon, Ciprofloxacin, Azithromycin und Doxycyclin, die nur noch empfindlich gegen Ertapenem und vermutlich Aminoglykoside sind12,13, könnten so innerhalb weniger Tage identifiziert werden.
Resistenz-Surveillance muss vergütet werden!
Sexuell übertragbare Infektionen nehmen zu. Auch das Risiko von antimikrobiellen Resistenzen bei sexuell übertragbaren Erregern nimmt zu, insbesondere durch Interventionen, wie Doxy-PEP. Angesichts dieser Entwicklung ist eine entsprechende Resistenz-Surveillance notwendig. Neisseria gonorrhoeae, Chlamydia trachomatis und Mycoplasma genitalium sind in der klassischen Mikrobiologie allerdings schwierig zu bearbeiten. Sie lassen sich nicht so gut anzüchten wie viele andere infektiöse Bakterien. Mittlerweile gibt es aber neue molekulardiagnostische Verfahren, die sich zum Nachweis von antimikrobieller Resistenz bei diesen sexuell übertragbaren Keimen eignen. Für die relevanten Antibiotika sind auch Daten zur Interpretation der Ergebnisse vorhanden. Eine breite Resistenz-Surveillance wäre somit möglich. Doch es scheitert an den Kosten. Trotz Meldepflicht von Resistenzen bzw. von schlecht ansprechenden LGV-assoziierten CT-Stämmen, wird die molekulardiagnostische Resistenztestung weder von Krankenkassen noch von anderen Kostenträgern vergütet. So bleiben in Deutschland aktuell nur Sentinel-Untersuchungen oder verschiedene Studienprojekte, die einen Einblick in die Epidemiologie geben.
Mycoplasma genitalium
Die Anzucht von Mycoplasma genitalium ist nur in Referenzlaboren möglich. Es gibt mittlerweile aber auch, neben diversen publizierten Methoden, mehrere kommerziell verfügbare Punktmutations-Assays, mit denen man eine Resistenz gegen Makrolid- und Fluorchinolon-Antibiotika detektieren kann.14 Die Analytik der 23S ribosomalen RNA für Makrolid-Antibiotika liefert zuverlässige Ergebnisse und die Übereinstimmung mit dem klinischen Ansprechen ist sehr gut. Bei Fluorchinolon-Antibiotika ist dagegen der negative prädiktive Wert deutlich besser als der positive prädiktive Wert. So kann trotz Nachweis von Mutationen im parC-Gen eine Therapie erfolgreich sein.15 Mutationen in der 16S ribosomalen RNA zeigen eine Assoziation mit Resistenz gegen Doxycyclin. Da sich hier bisher keine einzelne typische Mutation gezeigt hat (möglicher Kandidat: C1192T) ist die Sequenzierung des relevanten Genbereichs die Methode der Wahl.
Abhängig von der untersuchten Population (MSM in Berlin >80% Resistenz gegen Makrolide) ist vor Therapie eine Resistenzanalytik zu empfehlen. Die entsprechenden Tests sind verfügbar, werden aber in Deutschland nur von wenigen Labore angeboten, da es keine kostendeckende Vergütung durch die Krankenkassen gibt.
Chlamydia trachomatis
Mittel der Wahl gegen Chlamydia trachomatis ist Doxycyclin. Eine Resistenz gegen dieses Antibiotikum ist sehr selten und zudem nicht gut verstanden. Von einer Resistenz zu unterscheiden sind verschiedene CT-Varianten wie die Serovare L1-L3, die als Verursacher des Lymphogranuloma venereum eine längere Therapiedauer benötigen. Sie zeigen in der Zellkultur keinen höheren Resistenzgrad als andere Varianten.
Der Nachweis von tet-Genen, die wie tetM von anderen Bakterien im Genitalbereich (z.B. Lactobazillen) horizontal übertragen werden, oder wie tetC von Chlamydia suis16 übertragen wird, spielt bisher keine größere Rolle, auch wenn eine Assoziation mit schlechterem Therapieansprechen beschrieben ist.17 Der Nachweis von tetC aus Primärmaterial könnte stark prädiktiv für eine Resistenz sein, dennoch macht der tetM-Nachweis nur aus Reinkulturen Sinn, da dieses Gen in vielen anderen Bakterien der lokalen Flora nachgewiesen werden kann. Ferner gibt es auch Mutationen in der 23S ribosomalen RNA, die mit einer Resistenz gegen Doxycyclin, aber vor allem mit einer Resistenz gegen Azithromycin assoziiert sind. Mutationen im GyrA sowie im parC-Gen zeigen eine Assoziation mit Resistenz gegen Fluorchinolone bei bisher schlechter Datenlage.18
1 Brendefur Corwin, L., Campbell, P., Jakobsen, K., Müller, F., Lai, X., Unemo, M., Leegaard, T., Vildershøj Bjørnholt, J., and Olsen, A. (2023). Improvement in Neisseria gonorrhoeae culture rates by bedside inoculation and incubation at a clinic for sexually transmitted infections. Ann. Clin. Microbiol. Antimicrob. 22, 27. https://doi.org/10.1186/s12941-023-00576-0.
2 Pitt, R., Boampong, D., Day, M., Jensen, J.S., and Cole, M. (2022). Challenges of in vitro propagation and antimicrobial susceptibility testing of Mycoplasma genitalium. J. Antimicrob. Chemother. 77, 2901-2907. https://doi.org/10.1093/jac/dkac281.
3 Vogels, W.H., van Voorst Vader, P.C., and Schröder, F.P. (1993). Chlamydia trachomatis infection in a high-risk population: comparison of polymerase chain reaction and cell culture for diagnosis and follow-up. J. Clin. Microbiol. 31, 1103-1107.
4 Ja, G., Ts, P., R, P., S, B., V, C., and G, M. (2003). Evaluation of diagnostic efficacy of PCR methods for Chlamydia trachomatis infection in genital and urine specimens of symptomatic men and women in India. Jpn. J. Infect. Dis. 56.
5 Trembizki, E., Guy, R., Donovan, B., Kaldor, J.M., Lahra, M.M., and Whiley, D.M. (2016). Further evidence to support the individualised treatment of gonorrhoea with ciprofloxacin. Lancet Infect. Dis. 16, 1005–1006. https://doi.org/10.1016/S1473-3099(16)30271-7.
6 Szili, P., Draskovits, G., Révész, T., Bogár, F., Balogh, D., Martinek, T., Daruka, L., Spohn, R., Vásárhelyi, B.M., Czikkely, M., et al. (2019). Rapid Evolution of Reduced Susceptibility against a Balanced Dual-Targeting Antibiotic through Stepping-Stone Mutations. Antimicrob. Agents Chemother. 63, e00207-19. https://doi.org/10.1128/AAC.00207-19.
7 Rubin, D.H., Mortimer, T.D., and Grad, Y.H. (2023). Neisseria gonorrhoeae diagnostic escape from a gyrA-based test for ciprofloxacin susceptibility and the effect on zoliflodacin resistance: a bacterial genetics and experimental evolution study. Lancet Microbe 4, e247-e254. https://doi.org/10.1016/S2666-5247(22)00356-1.
8 Unemo, M., Seifert, H.S., Hook, E.W., Hawkes, S., Ndowa, F., and Dillon, J.-A.R. (2019). Gonorrhoea. Nat. Rev. Dis. Primer 5, 1-23. https://doi.org/10.1038/s41572-019-0128-6.
9 Mortimer, T.D., Zhang, J.J., Ma, K.C., and Grad, Y.H. (2022). Loci for prediction of penicillin and tetracycline susceptibility in Neisseria gonorrhoeae: a genome-wide association study. Lancet Microbe 3, e376-e381. https://doi.org/10.1016/S2666-5247 (22)00034-9.
10 Reichert, E., and Grad, Y.H. (2024). Effects of doxycycline post-exposure prophylaxis for prevention of sexually transmitted infections on gonorrhoea prevalence and antimicrobial resistance among men who have sex with men in the USA: a modelling study. Lancet Microbe 0. https://doi.org/10.1016/S2666-5247(24)00168-X.
11 Golparian, D., Sánchez-Busó, L., Cole, M., and Unemo, M. (2021). Neisseria gonorrhoeae Sequence Typing for Antimicrobial Resistance (NG-STAR) clonal complexes are consistent with genomic phylogeny and provide simple nomenclature, rapid visualization and antimicrobial resistance (AMR) lineage predictions. J. Antimicrob. Chemother. 76, 940-944. https://doi.org/10.1093/jac/dkaa552.
12 Hal, S.J. van, Sherry, N., Coombs, G., Mowlaboccus, S., Whiley, D.M., and Lahra, M.M. (2024). Emergence of an extensively drug-resistant Neisseria gonorrhoeae clone. Lancet Infect. Dis. 24, e547-e548. https://doi.org/10.1016/S1473-3099(24)00486-9.
13 Caméléna, F., Mérimèche, M., Brousseau, J., Mainardis, M., Verger, P., Le Risbé, C., Brottet, E., Thabuis, A., Bébéar, C., Molina, J.-M., et al. (2024). Emergence of Extensively Drug-Resistant Neisseria gonorrhoeae, France, 2023. Emerg. Infect. Dis. 30. https://doi.org/10.3201/eid3009.240557.
14 Le Roy, C., Bébéar, C., and Pereyre, S. Performance of Three Commercial Molecular Diagnostic Assays for the Simultaneous Detection of Mycoplasma genitalium and Macrolide Resistance. J. Clin. Microbiol. 59, e00020-21. https://doi.org/10.1128/JCM.00020-21.
15 Murray, G.L., Plummer, E.L., Bodiyabadu, K., Vodstr-cil, L.A., Huaman, J.L., Danielewski, J.A., Chua, T.P., Machalek, D.A., Garland, S., Doyle, M., et al. (2023). gyrA Mutations in Mycoplasma genitalium and Their Contribution to Moxifloxacin Failure: Time for the Next Generation of Resistance-Guided Therapy. Clin. Infect. Dis. Off. Publ. Infect. Dis. Soc. Am. 76, 2187-2195. https://doi.org/10.1093/cid/ciad057.
16 Marti, H., Suchland, R.J., and Rockey, D.D. (2022). The Impact of Lateral Gene Transfer in Chlamydia. Front. Cell. Infect. Microbiol. 12, 861899. https://doi.org/10.3389/fcimb.2022.861899.
17 Shao, L., You, C., Cao, J., Jiang, Y., Liu, Y., and Liu, Q. (2020). High treatment failure rate is better explained by resistance gene detection than by minimum inhibitory concentration in patients with urogenital Chlamydia trachomatis infection. Int. J. Infect. Dis. 96, 121-127. https://doi.org/10.1016/j.ijid.2020.03.015.
18 Benamri, I., Azzouzi, M., Sanak, K., Moussa, A., and Radouani, F. (2021). An overview of genes and mutations associated with Chlamydiae species’ resistance to antibiotics. Ann. Clin. Microbiol. Antimicrob. 20, 59. https://doi.org/10.1186/s12941-021-00465-4.









 Diese Seite weiter empfehlen
Diese Seite weiter empfehlen